Um alle Funktionen dieser Seite zu nutzen, aktivieren Sie bitte die Cookies in Ihrem Browser.
my.chemie.de
Mit einem my.chemie.de-Account haben Sie immer alles im Überblick - und können sich Ihre eigene Website und Ihren individuellen Newsletter konfigurieren.
- Meine Merkliste
- Meine gespeicherte Suche
- Meine gespeicherten Themen
- Meine Newsletter
Moleküldynamik
Weiteres empfehlenswertes FachwissenDer Begriff Moleküldynamik wird manchmal auch als Synonym für die Discrete element method (DEM) gebraucht, weil die Methoden sehr ähnlich sind. Die Partikel in DEM müssen aber keine chemische Moleküle sein. In den meisten Kontexten steht der Begriff MD für die Simulation im Bereich physikalischer Chemie. Physikalische PrinzipienMikrokanonisches Ensemble (NVE)Das System ist isoliert und tauscht keine Masse (N), Volumen (V) oder Energie (E) mit der Umgebung aus. Für ein System mit N Partikeln, mit zugehörigen Koordinaten X und Geschwindigkeiten Die Funktion der potenziellen Energie U(X) beschreibt dabei die gegenseitigen Anziehungen und Abstossungen der Moleküle. U(X) ist auch als Kraftfeld bekannt. Man erhält dieses meistens aus quantenmechanischen Berechnungen (Quantenchemie) und/oder spektroskopischen Experimenten. Trotzdem wird das Kraftfeld meist mit einer angepassten klassischen Funktion dargestellt. |
| Dieser Artikel basiert auf dem Artikel Moleküldynamik aus der freien Enzyklopädie Wikipedia und steht unter der GNU-Lizenz für freie Dokumentation. In der Wikipedia ist eine Liste der Autoren verfügbar. |





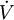 , kann man folgendes Paar gewöhnlicher Differentialgleichungen aufstellen:
, kann man folgendes Paar gewöhnlicher Differentialgleichungen aufstellen: